Studie: Wie entstand Omikron?

Wir finden die Entstehungsgeschichte von Omikron sehr spannend. Gleichzeitig können daraus möglicherweise wichtige Erkenntnisse für die Zukunft gewonnen werden. Daher schauen wir uns das Ganze nochmal genauer an:
Eine Studie[1] hat Omikron genauer unter die Lupe genommen. Sie hatte das Ziel, die wahrscheinlichste Wirtsart von Omikron zu bestimmen. Sie ist die Quelle für den folgenden Text und die Zitate.
Zur Erinnerung noch einmal die gängigen Theorien, wo sich die Omikron-Variante entwickelt haben könnte:
- In der Bevölkerung einer Region, in der es keine ausreichende Virusüberwachung gibt.
- Während einer anhaltenden (chronischen) COVID-19-Infektion in einem immungeschwächten Menschen.
- In einem nichtmenschlichen Wirt.
Spricht etwas gegen eine Entstehung von Omikron im Menschen?
Es gibt sogenannte „de-novo Mutationen” - neu entstandene Mutationen, die nicht während der Vervielfältigung des RNA-Strangs auftreten, sondern durch chemische Veränderungen an einzelnen Bausteinen innerhalb der RNA. Diese Art der Mutation hängt stark von der Umgebung ab, auf die ein Virus auf zellulärer Ebene trifft. Und diese genaue Zusammensetzung einer Zelle ist typisch für den Wirt. Viren neigen also in verschiedenen Wirtsarten zu unterschiedlichen Mutationstypen. Ein und dieselbe Virusart entwickelt sich also in unterschiedlichen Tierarten, auf der molekularen Ebene, unterschiedlich weiter. Dagegen werden selbst unterschiedliche Viren wie Polio- Ebola- und SARS-CoV-2-Viren, wenn sie sich in derselben Tierart entwickeln, auf ähnliche Weise verändert.
Die Forschenden machten sich also daran, durch die Analyse der Mutationen die Wirtsart zu bestimmen, in der Omikron entstanden sein kann: Sie identifizierten dafür die Mutationen, die Omikron vor seiner ersten Entdeckung in Südafrika erworben hatte und testeten zunächst, ob die Art der Mutationen mit der zellulären Umgebung menschlicher Wirte übereinstimmte. Dabei wurden zwischen Omikron und anderen Varianten, die sich bekanntermaßen im Menschen entwickelt haben, deutliche Unterschiede festgestellt. Unter den anderen Varianten waren auch 3, die sich während chronischer COVID-19-Infektionen gebildet hatten. Die Ergebnisse sprachen also gegen beide Theorien, die von einer Entstehung von Omikron im Menschen ausgehen.
Daraufhin verglichen die Forschenden Mutationen an diversen Viren, aus einer Vielzahl von Wirtssäugetieren, mit denen in Omikron. Die Studie bietet somit Einblicke in die Entwicklung dieser Virusvariante. Darüber hinaus schlagen die Forschenden Strategien vor, um zukünftige Ausbrüche von Varianten aus tierischen Zwischenwirten zu vermeiden.
„Die Art der Omikron-Mutationen, die sich vor der ersten Entdeckung in Südafrika entwickelt hatten, passt nicht zu einer Evolution im Menschen.”
Changshuo Weiab, Ke-Jia Shanab, Weiguang Wang et al. (Sinngemäß aus dem Englischen übersetzt.)
Gibt es Hinweise auf einen Wirtssprung?
Die identifizierten Mutationen und Vergleiche mit anderen Varianten legen nahe, dass der Vorfahre von Omikron etwa Mitte 2020 von anderen genetisch verwandten Varianten der B.1.1-Linie getrennt wurde. Im weiteren Verlauf, bis zum Ausbruch in Südafrika, hatte Omikron auffällig viele Mutationen erworben, die das Spike-Protein betreffen (Mutationen im ORF S). Omikron hatte hier, im Vergleich mit anderen Varianten, etwa 3,3 mal so schnell Mutationen angesammelt: durchschnittlich sind es bei SARS-CoV-2 etwa 0,45 Mutationen pro Monat, bei Omikron 1,5! Insbesondere die Region, die für die Bindung des Virus an die Wirtszelle zuständig ist (die kodierende Region der rezeptorbindenden Domäne (RBD)), wies viele Mutationen auf. Währenddessen war die Geschwindigkeit der Entstehung neuer Mutationen in anderen Bereichen Omikrons nicht erhöht.
Die Forschungsgruppe machte sich nun daran, den Grund für die schnelle Veränderung des Spike-Proteins aufzuzeigen:
Während der 18 Monate (Mai 2020 bis November 2021) hatten sich 45 Punktmutationen in Omikron angesammelt, von denen allein 27 das Spike-Protein betreffen. Die Forschenden fanden heraus, dass es sich bei 26 dieser 27 um „nicht synonyme Mutationen“ handelt. Dies weist darauf hin, dass eine starke „positive Selektion“[2] zu dieser auffälligen Veränderung beigetragen hatte: Das Spike-Protein der Omikron-Variante war offenbar unter einem großen Druck gewesen, sich an veränderte Bedingungen anzupassen. Keine der anderen bekannten Varianten, die sich im Menschen entwickelt hatten, war einem solchen Selektionsdruck ausgesetzt gewesen. Da das Spike-Protein bestimmt, welche Organismen (Tierarten) ein Coronavirus infizieren kann, war dies für die Forschenden ein weiterer Hinweis darauf, dass der Vorläufer von Omikron vom Menschen auf eine nichtmenschliche Art gesprungen sein könnte. Denn ein solcher Sprung würde zur schnellen Anpassung an den neuen Wirt erhebliche Mutationen im Spike-Protein erfordern. Währenddessen herrschen bei Varianten, die nur zwischen Menschen ausgetauscht werden, mehr oder weniger gleiche Verhältnisse in den Wirtszellen - und das Virus ist nicht auf solch eine starke Veränderung im Spike-Protein angewiesen.
Wie lässt sich der Wirt, in dem Omikron wahrscheinlich entstand, ermitteln?
Die ersten Erkenntnisse der Studie wiesen darauf hin, dass Omikron sich möglicherweise nicht im Menschen entwickelt hat. Nun versuchte die Forschungsgruppe, die nichtmenschliche Wirtsart zu bestimmen, in der sich vor dem Ausbruch in Südafrika die vielen Mutationen angesammelt haben können. Sie untersuchten Sequenzen von Hepatitisviren aus Mäusen, sowie Coronaviren, die sich in verschiedenen Säugetierarten entwickelt haben. Dabei identifizierten sie die jeweiligen Mutationen, die sich angesammelt hatten. Anschließend analysierten sie diese und weitere Mutationen. Darunter waren auch Mutationen menschlicher SARS-CoV-2-Varianten, sowie MERS-CoV-Mutationen, die sich im Menschen entwickelt hatten. Um das Ganze zu visualisieren, wurden die verschiedenen Virusvarianten anhand der ermittelten zwei Hauptkomponenten als Punkte in ein Koordinatensystem übertragen. Je nach Wirtsart, wurden sie unterschiedlich eingefärbt.
Wie sich zeigte, gruppierten sich die Varianten (bzw. die molekulare Bandbreite der Mutationen) nach den Wirtsarten. So konnten um die verschiedenen Wirtsarten sogenannte 95%-Vertrauensellipsen gezogen werden. Die unterschiedlichen Varianten, selbst die verschiedener Viren, hatten sich also in den jeweiligen Tierarten ähnlich entwickelt. Wahrscheinlich, weil die Viren Einflüssen (Mutagenen) ausgesetzt gewesen waren, die für die zelluläre Umgebung des jeweiligen Wirtes spezifisch sind und die entsprechenden Mutationen auslösten.
Die Ergebnisse decken sich damit, dass die Omikron-Mutationen, die sich nach der Entdeckung Omikrons entwickelten, im Menschen entstanden. Die vor dem Ausbruch in Südafrika entstandenen Mutationen scheinen sich dagegen in einem Nagetier entwickelt zu haben: ihre molekulare Bandbreite liegt innerhalb der Maus-Ellipse.
„Die Art der Omikron-Mutationen, die sich vor der ersten Entdeckung in Südafrika entwickelt hatten, stimmen mit einer Evolution in Mäusen überein.“ Changshuo Weiab, Ke-Jia Shanab, Weiguang Wang et al. (Sinngemäß aus dem Englischen übersetzt.)
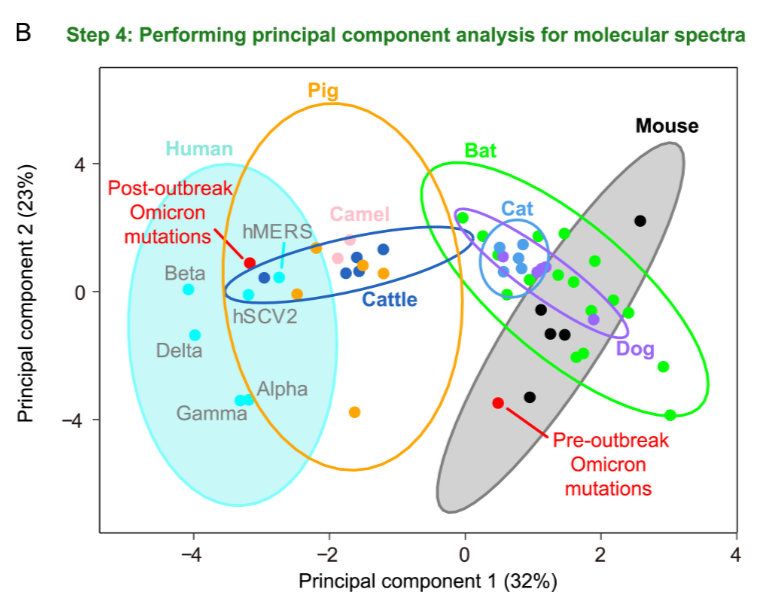
Step 4: Performing principal component analysis for molecular spectra (Ausschnitt aus dieser Datei (ext. Link))
*© 2021 The Authors. Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, and Genetics Society of China. Published by Elsevier Limited and Science Press.
„Zusammenfassend legen unsere Ergebnisse nahe, dass der Vorläufer von Omikron von Menschen auf Mäuse sprang, schnell Mutationen anhäufte die der Infektion dieses Wirts förderlich waren, und dann wieder auf Menschen zurück sprang. Dies weist für den Omikron-Ausbruch auf einen evolutionären Entwicklungsverlauf zwischen den Arten hin.“
Changshuo Weiab, Ke-Jia Shanab, Weiguang Wang et al. (Übersetzt aus dem Englischen)
Kann Omikron wirklich in Mäusen entstanden sein?
Kann Omikron tatsächlich in einer Tierart entstanden sein, die in Studien mit dem ursprünglichen SARS-2-Virus nicht infiziert werden konnte? Wenn ja - müssen wir mit weiteren Varianten aus Tierarten rechnen, die bisher nicht als Überträger gelten?
Ursprünglich galten Mäuse als schlechte Wirte für SARS-CoV-2. Das Spike-Protein der frühen Varianten war nicht an sie angepasst. Im Laufe der Pandemie tauchten jedoch Varianten auf, die Mäuse infizieren konnten. Wenn sich der Vorläufer von Omikron in einer Mausart entwickelt hat, müsste sich das Spike-Protein wahrscheinlich durch eine verbesserte Bindungsfähigkeit an das Enzym ACE2 (Angiotensin Converting Enzyme 2)[3] von Mäusen angepasst haben. Denn ACE2 dient einigen Coronaviren, darunter SARS-CoV-2, als Rezeptor und somit als Eintrittspforte in die Zellen des Wirtes: Die Spike-Proteine der Viren binden an das Enzym. Durch anschließende Fusion (Verschmelzung), gelangen die Viren ins Zellinnere, wo sie sich vermehren.
Untersuchungen der Forschungsgruppe ergaben, dass an Mäuse angepasste SARS-CoV-2-Varianten und die Omikron-Variante tatsächlich Mutationen teilen, die die Bindungsfähigkeit an ACE2 von Mäusen verbessern. Solche Veränderungen gab es weder in den anderen vier besorgniserregenden Varianten, noch in Varianten, die sich in chronisch infizierten Menschen bildeten. Dies stützt die vorangegangenen Untersuchungsergebnisse, nach denen sich Omikron wahrscheinlich in Mäusen entwickelte. Omikron könnte allerdings auch in einer anderen Art entstanden sein, die eine ähnliche „zelluläre Mutagenumgebung und ACE2-Struktur“ wie Mäuse aufweist. Die Omikron-Mutationen müssten die Bindungsfähigkeit an den ACE2-Rezeptor dieser Wirtsart verbessern.
Eine Überprüfung bestätigte, dass Mäuse, unter allen 32 getesteten Säugetieren, die wahrscheinlichste Wirtsart für den Vorläufer von Omikron sind. Die Untersuchungen („molekulare Docking-basierte Vorhersagen“) zeigten allerdings auch, dass die Anpassung von Omikron an Mäuse auch die Anpassung an andere Arten wie Menschen, Kamele und Ziegen gefördert hat. Dieser Effekt kommt vermutlich durch die strukturelle Ähnlichkeit des Enzyms ACE2 dieser Arten zustande. Das weist auf folgendes hin:
Sobald eine SARS-CoV-2-Variante die Fähigkeit erlangt, einen neuen Wirt zu infizieren, kann sie in diesem neuen tierischen Reservoir Mutationen ansammeln und wird auch übertragbarer auf andere Arten. Diese „Kettenreaktion“ des Sprungs von einem Wirt auf den anderen, könnte möglicherweise zu einer bemerkenswert hohen Vielfalt bei der Anpassung an die ACE2-Rezeptoren verschiedener Tierarten führen.
„Angesichts der Fähigkeit von SARS-CoV-2 auf verschiedene Arten zu springen, erscheint es wahrscheinlich, dass die Weltbevölkerung mit zusätzlichen tierischen Varianten konfrontiert sein wird, bis die Pandemie gut unter Kontrolle ist.“ Changshuo Weiab, Ke-Jia Shanab, Weiguang Wang et al. (Übersetzt aus dem Englischen.)[4]
Steigt die Gefahr der Verbreitung des Virus unter „Nutztieren”?
Und was können wir - die Bevölkerung - tun?
* Evidence for a mouse origin of the SARS-CoV-2 Omicron variant (Link zur Studie)
Author: Changshuo Wei,Ke-Jia Shan,Weiguang Wang,Shuya Zhang,Qing Huan,Wenfeng Qian
Publication: Journal of Genetics and Genomics
Publisher: Elsevier
Date: December 2021 ↩
Quellen:
Evidence for a mouse origin of the SARS-CoV-2 Omicron variant. Changshuo Weiab, Ke-Jia Shanab, Weiguang Wang et al.; 12.2021 (Science Direct) ↩︎
Selektion (Evolution) - Selektion auf Genebene. (Wikipedia) ↩︎
Angiotensin Converting Enzyme 2. Dr. Frank Antwerpes, Bijan Fink, Michael Wolf (DocCheck Flexicon) ↩︎
Evidence for a mouse origin of the SARS-CoV-2 Omicron variant. Changshuo Weiab, Ke-Jia Shanab, Weiguang Wang et al.; 12.2021 (Science Direct) ↩︎